实验室动态
研究室2021级硕士生陈聪在《Food Research International》发表研究论文

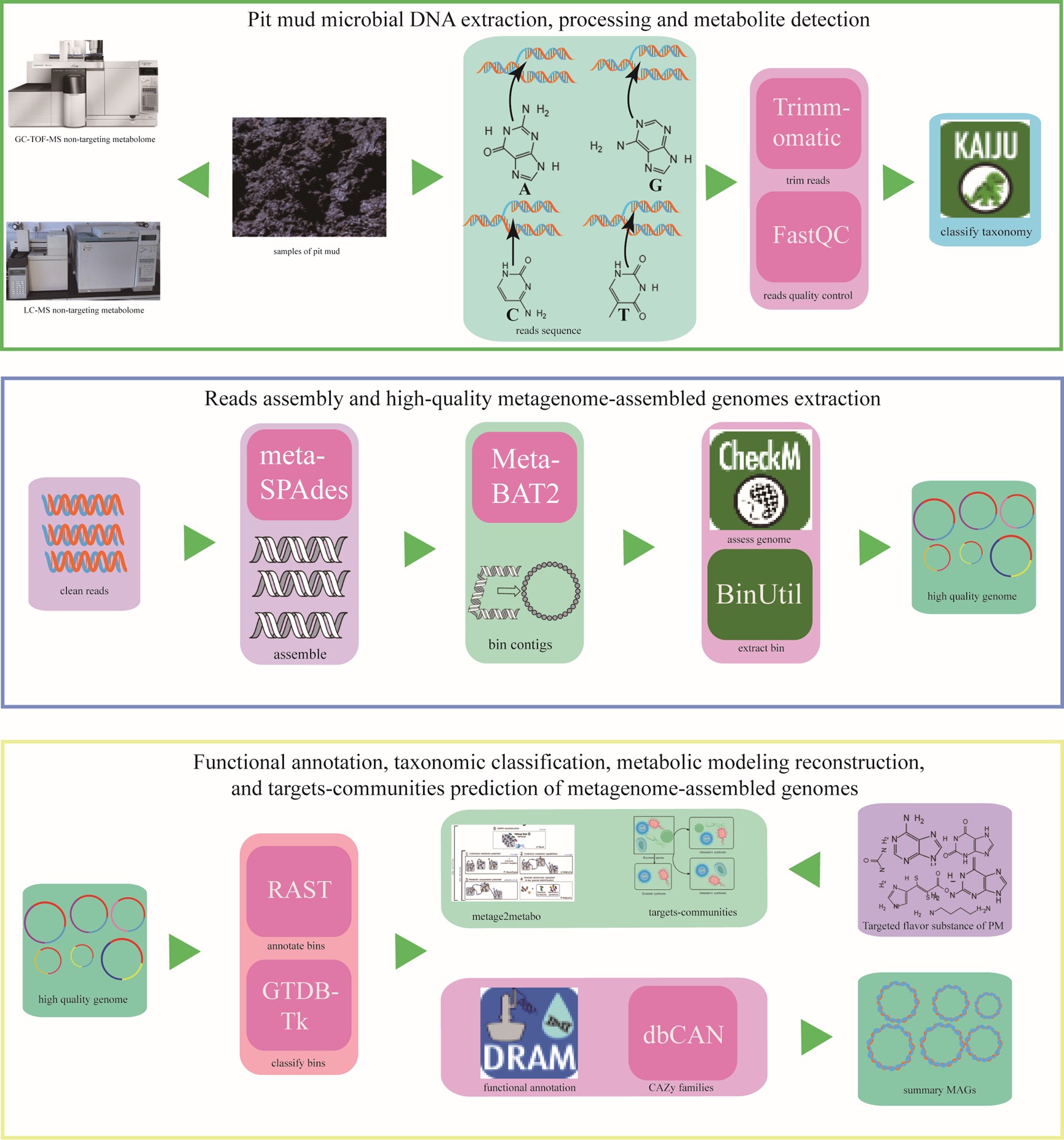
近日,白酒资源微生物与大数据应用课题组在农林科学领域1区TOP期刊《Food Research International》 (Q1, IF:8.1)发表题为“Revealing microbiota characteristics and predicting flavor-producing sub-communities in Nongxiangxing baijiu pit mud through metagenomic analysis and metabolic modeling”的研究性论文(https://doi.org/10.1016/j.foodres.2024.114507)。四川轻化工大学2021级硕士研究生陈聪为论文第一作者,邹伟副教授为论文通讯作者。该研究工作得到了四川省自然科学基金面上项目(2023NSFSC0184)的支持。 白酒是中国国酒。窖泥微生物群落对浓香型白酒风味物质形成具有重要影响。如何通过解析窖泥微生物群落的结构和功能来阐释浓香型白酒关键风味物质形成机理是目前白酒研究的热点。由于存在大量难培养微生物以及缺乏窖泥微生物参考基因组,窖泥中微生物群落功能机制解析受到了极大地限制。本论文结合宏基因组技术和代谢网络模型分析,从窖泥宏基因组序列中组装基因组,并通过基因组测序与注释、结合相关生物信息数据、实验数据,构建窖泥微生物高质量基因组规模代谢网络模型(Genome scale metabolic model,GSMM)。同时,借助基于约束算法、网络扩展算法以及代谢组学数据分析窖泥微生物基本生理代谢特征,预测与窖泥风味物质合成相关的窖泥微生物和子群落。目前,本论文借助宏基因组学和代谢网络模型解析白酒生态系统微生物群落尚属首例。研究中共获得38个高质量的宏基因组组装基因组(MAGs),包括家族级(13个)、属级(17个)和种级(8个)。随后构建了这38个MAGs的GSMMs。通过GSMMs,分别预测个体和群落具有生产111种代谢物和598种代谢物的能力。其中23种预测代谢物与检测到的代谢组学风味一致,并被作为目标物质。通过38个GSMMs的互作模拟筛选出12个亚群落。在每个亚群落中,确定了Methanobacterium、Sphaerochaeta、Muricomes intestini、Methanobacteriaceae、Synergistaceae和Caloramator是目标物质的核心微生物。本论文中关于宏基因组和目标群落筛选有助于解析白酒代谢-微生物组的关联和进一步的生物调控策略设计。

